Eine Firma in Berlin-Adlershof hat eine Methode entwickelt, mit der Forscher gezielt Proteine aus einer Lösung fischen können.
Das gesamte menschliche Genom ist entschlüsselt, die Funktion der Genprodukte, also der Proteine, aber bei weitem nicht. Dabei sind sie Angriffspunkte für viele Medikamente. Die Adlershofer Firma caprotec hat eine verblüffend einfache Methode entwickelt, um Wechselwirkungen von Proteinen mit kleinen Molekülen zu untersuchen. Sie verwenden sogenannte Capture Compounds, mit denen sie Proteine aus einer Lösung herausangeln können wie Fische aus einem Teich. Was dem Angler selten gelingt, nämlich eine ganz bestimmte Fischart zu fangen, haben die Wissenschaftler von caprotec perfektioniert: Sie können ihre Köder so gestalten, dass diese nur bestimmten Proteinen „schmecken“. Die maßgeschneiderten Capture Compounds helfen Forschern und Pharmaunternehmen, Auslöser für Nebenwirkungen von Arzneistoffen zu finden oder neue Medikamente zu entwickeln. Wissenschaftler vom Leibniz-Institut für Molekulare Pharmakologie (FMP) unterstützen die Adlershofer im Rahmen eines kürzlich vom BMBF mit einer Forschungsprämie ausgezeichneten Kooperationsprojektes mit theoretischen Berechnungen für das Design der Capture Compounds.
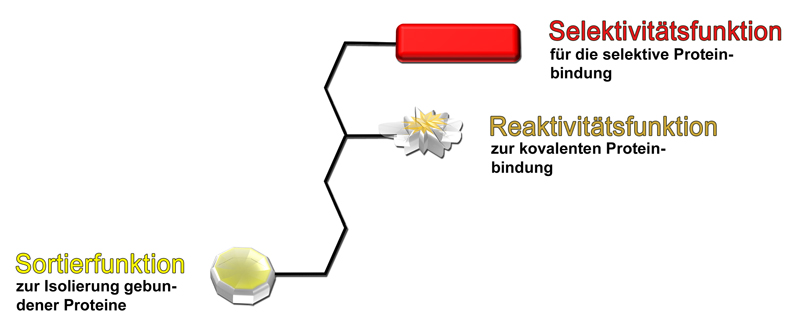
Capture Compounds bestehen aus drei Komponenten. Da ist zunächst der „Köder“, ein kleines Molekül, für dessen Eigenschaften sich die Forscher und Pharmaunternehmen interessieren. „Wir können dafür nahezu jedes Molekül verwenden, zum Beispiel einen neuen Wirkstoff“, sagt Dr. Christian Jurinke von caprotec. Passt ein solches Molekül in eine Bindungstasche eines oder mehrerer Proteine, docken die Proteine auf Grund von chemischen Wechselwirkungen locker an die Capture Compound an. In der Nähe dieses Köders befindet sich der „Haken“. Es ist eine funktionelle Gruppe, die beim Bestrahlen mit UV-Licht eine feste – sogenannte kovalente – Bindung mit dem angezogenen Protein eingeht: Es hängt nun fest an der Angel. Am anderen Ende der Capture Compound gibt es eine Sortierfunktion (Biotin). Diese regiert mit Streptavidin, das wiederum auf magnetische Kügelchen aufgebracht ist. Die Kügelchen lassen sich nun einfach aus einer Lösung von Proteinen herausfiltern. Die Zusammensetzung der Proteine, etwa in einer aufgeschlossenen Gewebeprobe, analysieren die Forscher schließlich per Massenspektrometer.
Was recht einfach klingt, erfordert eine ausgefeilte Entwicklungsarbeit und hier kommen Dr. Roland Kühne und Dr. Anna Schrey der Arbeitsgruppe Molecular Modeling vom FMP ins Spiel. Sie berechnen am Computer die Struktur der Bindungstaschen und können so beispielsweise vorhersagen, mit welcher Seite sich das kleine Ködermolekül in die Proteintasche schmiegt. Die Entwickler von caprotec wissen dann, wie herum sie das Molekül an die Capture Compound anbinden müssen.
Kontakt:
|
Dr. Christian Jurinke caprotec bioanalytics GmbH Volmerstr. 5, 12489 Berlin Tel.: 030 6392 3992 E-Mail: christian.jurinkecaprotec.com www.caprotec.com
Dr. Ronald Kühne |